Extraire et nettoyer des données bibliométriques avec R (1)
Exploring Scopus
Table des matières
Après le guide pour apprendre à programmer en R où j’ai listé des tutoriels et packages utiles, je propose ici d’entrer dans le vif du sujet et d’apprendre à extraire et nettoyer des données bibliométriques. Ce tutoriel garde à l’esprit les problèmes que peuvent rencontrer les historiens de l’économie, mais j’espère qu’il pourra également être utile à d’autres chercheurs en sciences sociales.
Il n’est pas facile de naviguer entre les différentes bases de données bibliométriques existantes (Web of Science, Scopus, Dimensions, Jstor, Econlit, RePEc…), et de savoir quelles informations elles collectent et quelle quantité de données on peut en extraire. J’apporterai donc ici une série de tutoriels sur la manière de trouver et d’extraire de telles données (si possible, en le faisant via des scripts R), mais aussi sur la manière de les nettoyer. En effet, selon la base de données utilisée, les données sont dans des formats différents, et il faut souvent faire un peu de nettoyage (notamment pour les références bibliographiques citées par le corpus que l’on a extrait). Dans ce tutoriel, nous nous concentrerons sur les données Scopus pour lesquelles vous aurez besoin d’un accès institutionnel. Le prochain post se concentre sur les données de Dimensions et le suivant traitera de l’application Constellate de JSTOR. Dimensions et Constellate sont accessibles sans accès institutionnel.
A la fin de ce tutoriel, vous saurez comment obtenir les données nécessaires pour construire des réseaux bibliographiques et je donnerai un exemple de réseau de co-citations en utilisant le package biblionetwork (Goutsmedt, Claveau, and Truc 2021). Ce dont vous avez seulement besoin dans ce tutoriel est une compréhension de base de certaines fonctions des paquets Tidyverse (Wickham 2021; Wickham et al. 2019)–principalement dplyr et stringr–ainsi qu’une compréhension (moins) basique de “regex” (Expressions Régulières)[^1]. À certains moments, je me plonge dans des méthodes plus compliquées et tortueuses en utilisant Elsevier/Scopus APIs avec rscopus ou en nettoyant des références avec le logiciel d’apprentissage automatique anystyle en utilisant la ligne de commande. Ceci sera expliqué dans des sections séparées que les débutants peuvent sauter.
[^1] : Ne vous inquiétez pas si vous ne comprenez pas les expressions régulières au début, c’est une bonne occasion d’apprendre et de pratiquer. Vous pouvez trouver des exemples plus simples ici et apprendre stringr par la même occasion.
Chargeons d’abord tous les paquets dont nous avons besoin et définissons le chemin vers le répertoire où je place les données extraites de Scopus :
# packages
package_list <- c(
"here", # use for paths creation
"tidyverse",
"bib2df", # for cleaning .bib data
"janitor", # useful functions for cleaning imported data
"rscopus", # using Scopus API
"biblionetwork", # creating edges
"tidygraph", # for creating networks
"ggraph" # plotting networks
)
for (p in package_list) {
if (p %in% installed.packages() == FALSE) {
install.packages(p, dependencies = TRUE)
}
library(p, character.only = TRUE)
}
github_list <- c(
"agoutsmedt/networkflow", # manipulating network
"ParkerICI/vite" # needed for the spatialisation of the network
)
for (p in github_list) {
if (gsub(".*/", "", p) %in% installed.packages() == FALSE) {
devtools::install_github(p)
}
library(gsub(".*/", "", p), character.only = TRUE)
}
# paths
data_path <- here(path.expand("~"),
"data",
"tuto_biblio_dsge")
scopus_path <- here(data_path,
"scopus")
Extraire des données de Scopus
Utilisation du site web de Scopus
Je me concentre ici sur de l’histoire récente : les modèles d’équilibre général stochastique dynamique (DGSE, en anglais). Ce type de modèles est apparu à la fin des années 1990 et est devenu la norme dans les publications universitaires ainsi que dans les institutions chargées de l’élaboration des politiques, notamment dans les banques centrales.1.
Si vous n’en avez pas, vous devrez créer un compte sur Scopus en utilisant votre adresse électronique institutionnelle. Une fois sur la page de recherche “Documents”, vous pouvez rechercher “DSGE” ou “Dynamic Stochastic General Equilibrium” dans les titres, les résumés et les mots-clés des documents (voir Figure 1). Le 20 janvier 2022, j’ai obtenu 2633 résultats. Vous pouvez sélectionner “Tous” les documents puis cliquer sur la flèche vers le bas à droite de “CSV export” (voir Figure 2). Vous devez alors choisir toutes les informations que vous souhaitez télécharger dans un fichier .csv. Ce dont nous avons besoin, c’est de cocher la case “Inclure les références” car nous en avons besoin pour la co-citation bibliographique plus tard (voir la figure 3)2.
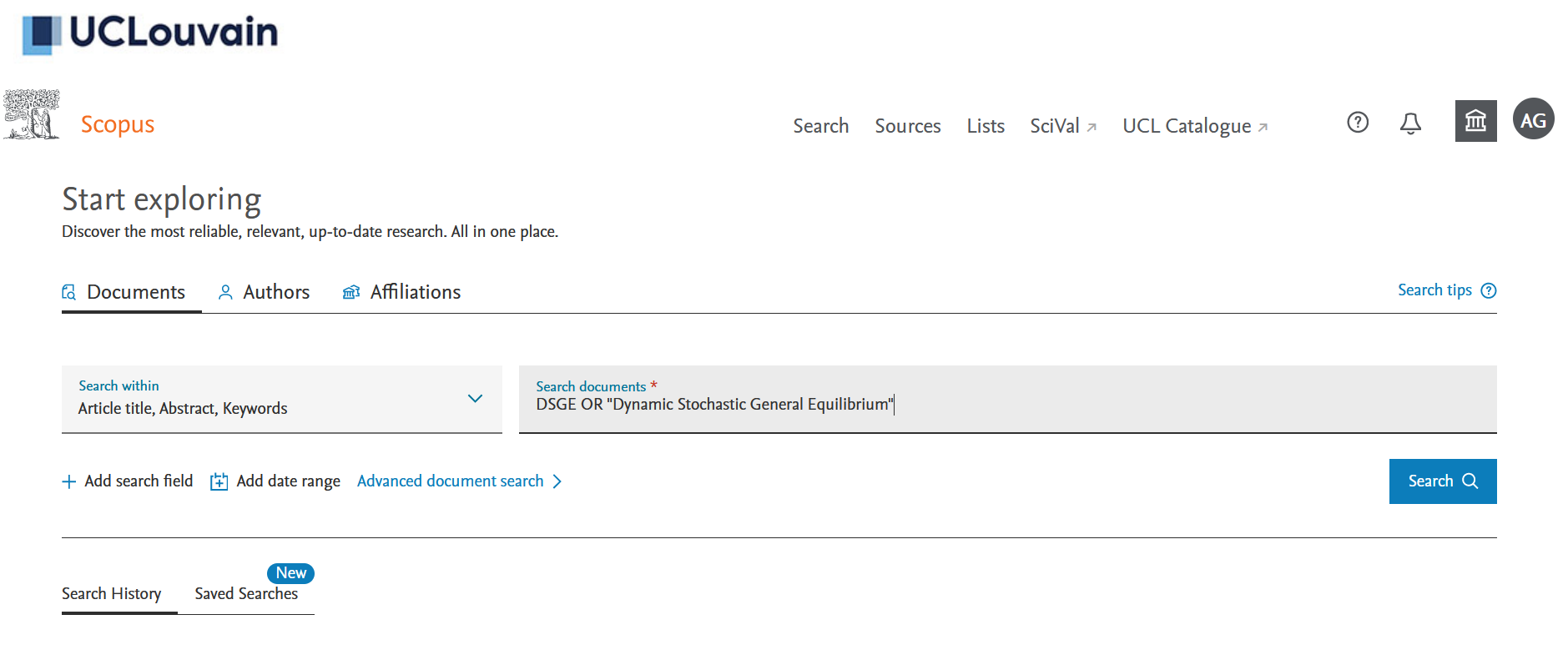
Figure 1: Scopus search page
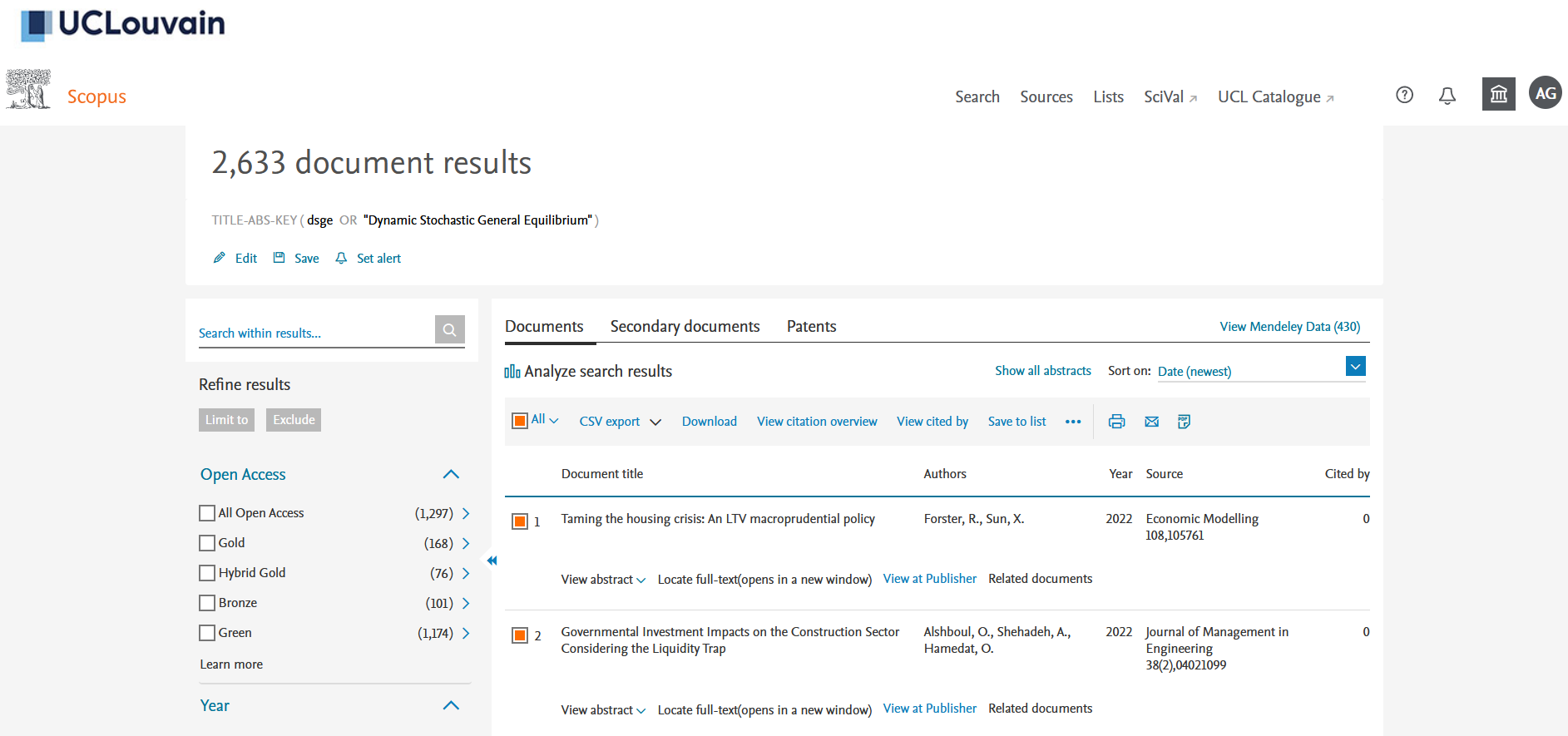
Figure 2: Scopus results page
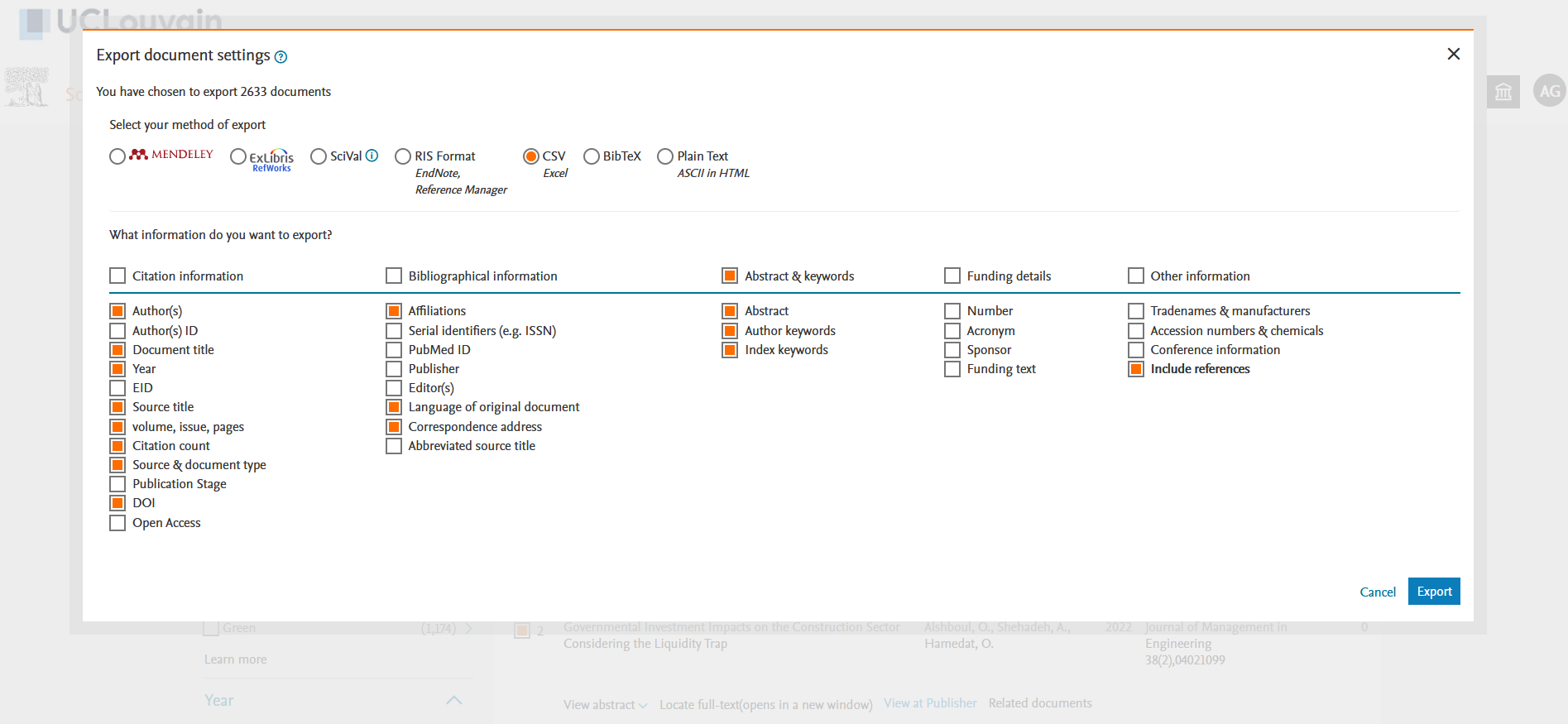
Figure 3: Choosing the information to export in a CSV
Le fichier .csv que vous avez exporté rassemble des métadonnées sur les auteurs, le titre, la revue, le résumé, les mots-clés, les affiliations et les références sur les documents (principalement des articles, mais aussi des documents de conférence, des chapitres de livres et des revues) mentionnant DSGE dans leur titre, leur résumé ou leurs mots-clés. Cette méthode d’extraction de données présente deux limites :
- la quantité de données que vous pouvez exporter est limitée à 2000, ce qui est peu ;
- une partie des métadonnées est relativement brute, comme les affiliations et les références, ce qui implique un certain nettoyage.
Je vous montrerai comment nettoyer ces données brutes dans la prochaine section. Mais la sous-section suivante vous explique comment utiliser l’API de Scopus directement dans R, pour interroger plus facilement différentes données et extraire plus d’éléments.
Méthode alternative : Utiliser les API de Scopus et rscopus
Vous devez d’abord créer une “clé API” sur le site Web de Scopus (voir plus d’informations ici) associée à votre compte. L’utilisation des APIs vous permet d’extraire un plus grand nombre de données (voir les API disponibles et les quotas correspondants ici).
Utilisons Rscopus et définissons la clé API :
api_key <- "your_api_key"
set_api_key(api_key)
La plupart du temps (et c’était le cas pour moi), votre accès institutionnel est lié à une adresse IP et vous ne pouvez pas utiliser les API si vous n’êtes pas connecté au réseau Internet de votre institution. Si vous souhaitez travailler à distance, vous devez demander une authentification “basée sur un jeton” et demander un “jeton institutionnel” ou “Insttoken” (voir les explications ici). Il vous suffit d’écrire à Elsevier (https://service.elsevier.com/app/contact/supporthub/researchproductsapis/) pour expliquer le type de recherche que vous menez et leur demander un “insttoken”. C’est aussi une bonne occasion, si nécessaire, de leur demander des quotas plus élevés.
Définissons le jeton institutionnel :
insttoken <- "your_institutional_token"
insttoken <- inst_token_header(insttoken)
Nous lançons d’abord la requête en utilisant “Scopus Search API” via rscopus. Il s’agit de l’API correspondant à la recherche que nous avons effectuée [ci-dessus] (#using-scopus-website) sur le site web de Scopus. Nous pouvons maintenant extraire directement jusqu’à 20000 articles par semaine. Nous obtenons des données brutes avec beaucoup d’informations (les données mais aussi des informations sur la requête, etc.) En utilisant la fonction rscopus gen_entries_to_df, nous convertissons ces données brutes en data.frames.
dsge_query <- rscopus::scopus_search("TITLE-ABS-KEY(DSGE) OR TITLE-ABS-KEY(\"Dynamic Stochastic General Equilibrium\")",
view = "COMPLETE",
headers = insttoken)
dsge_data_raw <- gen_entries_to_df(dsge_query$entries)
Nous pouvons enfin séparer les différents data.frames. Nous obtenons trois tableaux contenant différents types d’informations :
- Un tableau avec tous les articles et leurs métadonnées ;
- Un tableau avec la liste de tous les auteurs des articles ;
- Un tableau avec la liste des affiliations.
dsge_papers <- dsge_data_raw$df
dsge_affiliations <- dsge_data_raw$affiliation
dsge_authors <- dsge_data_raw$author
Maintenant que nous avons les articles, nous devons extraire les références en utilisant scopus “Abstract Retrieval API”. Nous utilisons l’identifiant interne des articles pour trouver les références. Mais nous ne pouvons pas interroger les références avec plusieurs identifiants, nous devons donc faire une boucle pour extraire les références une par une. Nous créons une liste où nous mettons les références pour chaque article (nous avons autant de data.frames que d’articles dans notre liste) et nous lions les data.frames ensemble, en associant chacun d’eux à l’identifiant de l’article citant correspondant.3.
[Modification du 16 novembre 2023, suite à un commentaire d’Olivier :] Un autre problème avec l’extraction des références est que vous ne pouvez pas extraire plus de 40 références par article. Si un article a plus de 40 références dans sa bibliographie, il faut faire une boucle pour collecter toutes les références.
citing_articles <- dsge_papers$`dc:identifier` # extracting the IDs of our articles on dsge
citation_list <- list()
for(i in 1:length(citing_articles)){
citations_query <- abstract_retrieval(citing_articles[i],
identifier = "scopus_id",
view = "REF",
headers = insttoken)
if(!is.null(citations_query$content$`abstracts-retrieval-response`)){ # Checking if the article has some references before collecting them
nb_ref <- as.numeric(citations_query$content$`abstracts-retrieval-response`$references$`@total-references`)
citations <- gen_entries_to_df(citations_query$content$`abstracts-retrieval-response`$references$reference)$df
if(nb_ref > 40){ # The loop to collect all the references
nb_query_left_to_do <- floor((nb_ref) / 40)
cat("Number of requests left to do :", nb_query_left_to_do, "\n")
for (j in 1:nb_query_left_to_do){
cat("Request n°", j , "\n")
citations_query <- abstract_retrieval(citing_articles[i],
identifier = "scopus_id",
view = "REF",
startref = 40*j+1,
headers = insttoken)
citations_sup <- gen_entries_to_df(citations_query$content$`abstracts-retrieval-response`$references$reference)$df
citations <- bind_rows(citations, citations_sup)
}
}
citations <- citations %>%
as_tibble(.name_repair = "unique") %>%
select_if(~!all(is.na(.)))
citation_list[[citing_articles[i]]] <- citations
}
}
dsge_references <- bind_rows(citation_list, .id = "citing_art")
Une fois que vous avez ces quatre data.frames (métadonnées des articles, auteurs, affiliations et références), vous pouvez procéder à l’analyse bibliométrique. La seule difficulté est maintenant de naviguer entre les nombreuses colonnes de chaque data.frame et de comprendre ce que sont les différentes informations. Pour relier les métadonnées du data.frame des articles à celles de la référence, vous pouvez utiliser l’identifiant scopus que nous avons placé dans la table de référence (dans la colonne citing_art). Si vous voulez joindre les métadonnées des articles avec celles des auteurs et des affiliations, vous devez utiliser la colonne entry_number qui existe dans les trois data.frames. Ce numéro est créé par Scopus après votre requête, ce qui signifie que les articles, les affiliations et les auteurs ne sont pas liés par un identifiant permanent. Par conséquent, les identifiants seront régénérés et donc différents si vous changez votre requête.
En plus de vous épargner la tâche laborieuse de nettoyer les références et les affiliations, lorsque vous utilisez les API, les références ont déjà un identifiant, donné par Scopus. Cela signifie que si deux articles citent la même référence, vous le saurez car cette référence aura un identifiant commun dans les citations du premier et du second article. Plus bas, lorsque nous manipulerons les données extraites du site web de Scopus, nous devrons trouver nous-mêmes quelles citations correspondent à la même référence.4.
Si vous n’avez pas peur des catégories et de la langue de Scopus, ou si vous interrogez un site web par le biais d’un script R, c’est peut-être la méthode la plus rapide pour obtenir des données (et c’est une méthode qui vous permet d’obtenir plus de données, avec des requêtes plus compliquées). Cependant, dans la section suivante, je vous montre comment nettoyer les données extraites du site web de Scopus ci-dessus.
Nettoyage des données Scopus
Revenons aux données que nous avons téléchargées sur le site Scopus :
#' # Cleaning scopus data from website search
#'
scopus_search_1 <- read_csv(here(scopus_path, "scopus_search_1998-2013.csv"))
scopus_search_2 <- read_csv(here(scopus_path, "scopus_search_2014-2021.csv"))
scopus_search <- rbind(scopus_search_1, scopus_search_2) %>%
mutate(citing_id = paste0("A", 1:n())) %>% # We create a unique ID for each doc of our corpus
clean_names() # janitor function to clean column names
scopus_search
## # A tibble: 2,608 × 23
## authors title year source_title volume issue art_no page_start page_end
## <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 Ha J., So I. Infl… 2013 Global Econ… 42 4 <NA> 396 424
## 2 Botero J., … Exog… 2013 Revista de … 16 1 <NA> 1 24
## 3 Kirsanova T… Comm… 2013 Internation… 9 4 <NA> 99 151
## 4 Ashimov A.A… Para… 2013 Economic De… <NA> <NA> <NA> 95 188
## 5 Khan A., Th… Cred… 2013 Journal of … 121 6 <NA> 1055 1107
## 6 Jeřábek T.,… Pred… 2013 Acta Univer… 61 7 <NA> 2229 2238
## 7 Hu X., Xu B… Infl… 2013 Journal of … 5 12 <NA> 636 641
## 8 Cha H. Taki… 2013 Internation… 7 2 <NA> 280 296
## 9 da Silva M.… The … 2013 North Ameri… 26 <NA> <NA> 266 281
## 10 Sandri D., … Fina… 2013 Journal of … 45 SUPP… <NA> 59 86
## # ℹ 2,598 more rows
## # ℹ 14 more variables: page_count <dbl>, cited_by <dbl>, doi <chr>,
## # affiliations <chr>, authors_with_affiliations <chr>, abstract <chr>,
## # author_keywords <chr>, index_keywords <chr>, references <chr>,
## # correspondence_address <chr>, language_of_original_document <chr>,
## # document_type <chr>, source <chr>, citing_id <chr>
Il y a plusieurs choses à nettoyer :
- Nous avons plusieurs “auteurs” par document. Pour certaines analyses, par exemple les réseaux de co-auteurs, il est préférable d’avoir une “table d’auteurs” qui associe chaque auteur à une liste d’articles ;
- Nous avons plusieurs
affiliationspar article ainsi que plusieursauthors_with_affiliations. Cela nous permet de relier les auteurs à leurs affiliations, mais là encore nous devons les séparer en autant de lignes que d’auteurs (si je ne me trompe pas, il semble qu’il n’y ait qu’une seule affiliation par auteur dans cet ensemble de données) ; references;- Peut-être pour séparer
author_keywordsetindex_keywordssi vous voulez les utiliser.
Dans ce qui suit, nous nettoyons scopus_search afin de produire 3 data.frames supplémentaires :
- un
data.framequi associe chaque article à une liste d’auteurs et leur affiliation correspondante (voir ci-dessous) ; - un data.frame qui associe chaque article à la liste des références qu’il cite (un article a autant de lignes que le nombre de références citées) : c’est une table de “citation directe” (voir ici) ;
- une liste de toutes les références citées, qui permet de retrouver les références identiques dans le tableau des citations directes (voir cette sous-section)
Extraction des affiliations et des auteurs
Nous avons deux colonnes pour les affiliations :
- une colonne
affiliationsavec les affiliations seules ; - une colonne,
authors_with_affiliationsavec les auteurs et les affiliations.
affiliations_raw <- scopus_search %>%
select(citing_id, authors, affiliations, authors_with_affiliations)
knitr::kable(head(affiliations_raw, n = 2))
| citing_id | authors | affiliations | authors_with_affiliations |
|---|---|---|---|
| A1 | Ha J., So I. | Department of Economics, Cornell University, Ithaca, NY, United States; Department of Economics, University of Washington, Seattle, WA, United States | Ha, J., Department of Economics, Cornell University, Ithaca, NY, United States; So, I., Department of Economics, University of Washington, Seattle, WA, United States |
| A2 | Botero J., Franco H., Hurtado Á., Mesa M. | Departamento de Economía, Universidad EAFIT, Medellín, Colombia; EAFIT University, Colombia | Botero, J., Departamento de Economía, Universidad EAFIT, Medellín, Colombia; Franco, H., Departamento de Economía, Universidad EAFIT, Medellín, Colombia; Hurtado, Á., Departamento de Economía, Universidad EAFIT, Medellín, Colombia; Mesa, M., EAFIT University, Colombia |
Pour un nettoyage plus sûr, nous optons pour la deuxième colonne, car elle nous permet d’associer l’auteur à sa propre affiliation, comme décrit dans la colonne authors_with_affiliations. La stratégie consiste ici à séparer chaque auteur dans la colonne authors, puis à séparer les différents auteurs et affiliations dans la colonne authors_with_affiliations. Enfin, nous ne conservons que les lignes où l’auteur de la colonne authors_with_affiliations est le même que celui de la colonne authors
scopus_affiliations <- affiliations_raw %>%
separate_rows(authors, sep = ", ") %>%
separate_rows(contains("with"), sep = "; ") %>%
mutate(authors_from_affiliation = str_extract(authors_with_affiliations,
"^(.+?)\\.(?=,)"),
authors_from_affiliation = str_remove(authors_from_affiliation, ","),
affiliations = str_remove(authors_with_affiliations, "^(.+?)\\., "),
country = str_remove(affiliations, ".*, ")) %>% # Country is after the last comma
filter(authors == authors_from_affiliation) %>%
select(citing_id, authors, affiliations, country)
knitr::kable(head(scopus_affiliations))
| citing_id | authors | affiliations | country |
|---|---|---|---|
| A1 | Ha J. | Department of Economics, Cornell University, Ithaca, NY, United States | United States |
| A1 | So I. | Department of Economics, University of Washington, Seattle, WA, United States | United States |
| A2 | Botero J. | Departamento de Economía, Universidad EAFIT, Medellín, Colombia | Colombia |
| A2 | Franco H. | Departamento de Economía, Universidad EAFIT, Medellín, Colombia | Colombia |
| A2 | Hurtado Á. | Departamento de Economía, Universidad EAFIT, Medellín, Colombia | Colombia |
| A2 | Mesa M. | EAFIT University, Colombia | Colombia |
Nettoyer les références
Commençons par extraire la colonne references en conservant l’identifiant de l’article citant. Nous mettons chaque référence citée par un article sur une ligne séparée, en utilisant le fait que les références sont séparées par un point-virgule. Nous créons un identifiant pour chaque référence.
#' ## Extracting and cleaning references
references_extract <- scopus_search %>%
filter(! is.na(references)) %>%
select(citing_id, references) %>%
separate_rows(references, sep = "; ") %>%
mutate(id_ref = 1:n()) %>%
as_tibble
knitr::kable(head(references_extract))
| citing_id | references | id_ref |
|---|---|---|
| A1 | Bernanke, B., Gertler, M., Gilchrist, S., Financial accelerator in a quantitative business cycle framework, NBER Working Paper No. W6455 (1998), NBER | 1 |
| A1 | Calvo, G., Staggered prices in a utility maximizing framework (1983) Journal of Monetary Economics, 12, pp. 383-398 | 2 |
| A1 | Christensen, I., Dib, A., Monetary policy in an estimated DSGE model with a financial accelerator (2005) Computing in Economics and Finance, p. 314 | 3 |
| A1 | Dixit, A., Stiglitz, J., Monopolistic competition and optimum product diversity (1977) American Economic Review, 67 (3), pp. 297-308 | 4 |
| A1 | Gerali, A., Neri, S., Sessa, L., Signoretti, F., Credit and banking in a DSGE model (2010), 42 (6), pp. 107-141. , Working paper, Banka D’Italia, Rome | 5 |
| A1 | Goodfriend, M., McCallum, B., Banking and interest rates in monetary policy analysis: A quantitative exploration (2007), NBER Working Paper Series No. 13207, NBER | 6 |
Nous avons maintenant une référence par ligne et les complications commencent. Nous devons trouver des moyens d’extraire les différents éléments d’information pour chaque référence : auteurs, année, pages, volume et numéro, journal, titre, etc… Voici quelques expressions rationnelles qui permettront d’extraire certaines de ces informations.
extract_authors <- ".*[:upper:][:alpha:]+( Jr(.)?)?, ([A-Z]\\.[ -]?)?([A-Z]\\.[ -]?)?([A-Z]\\.)?[A-Z]\\."
extract_year_brackets <- "(?<=\\()\\d{4}(?=\\))"
extract_pages <- "(?<= (p)?p\\. )([A-Z])?\\d+(-([A-Z])?\\d+)?"
extract_volume_and_number <- "(?<=( |^)?)\\d+ \\(\\d+(-\\d+)?\\)"
Nous pouvons maintenant extraire les auteurs et l’année. Nous créons une nouvelle colonne, remaining_ref qui conserve les informations de la colonne references mais nous en retirons les auteurs. Pour faciliter le nettoyage, nous séparons les références en fonction de la position de l’année de publication dans la référence. Nous utilisons la variable is_article pour déterminer où se trouve l’année et donc si le titre est avant l’année ou non.
cleaning_references <- references_extract %>%
mutate(authors = str_extract(references, paste0(extract_authors, "(?=, )")),
remaining_ref = str_remove(references, paste0(extract_authors, ", ")), # cleaning from authors
is_article = ! str_detect(remaining_ref, "^\\([:digit:]{4}"),
year = str_extract(references, extract_year_brackets) %>% as.integer)
Je ne peux pas détailler toutes les regex ci-dessous mais le but est d’extraire le plus de métadonnées pertinentes possible, d’abord pour les références dont l’année de publication suit le titre (is_article == TRUE), ce qui correspond la plupart du temps à des articles de journaux.
Ce code a été écrit en janvier 2022 et j’essaierai de l’améliorer plus tard, notamment parce que l’utilisation de la variable
is_articlerallonge inutilement le code.
cleaning_articles <- cleaning_references %>%
filter(is_article == TRUE) %>%
mutate(title = str_extract(remaining_ref, ".*(?=\\(\\d{4})"), # pre date extraction
journal_to_clean = str_extract(remaining_ref, "(?<=\\d{4}\\)).*"), # post date extraction
journal_to_clean = str_remove(journal_to_clean, "^,") %>% str_trim("both"), # cleaning a bit the journal info column
pages = str_extract(journal_to_clean, extract_pages), # extracting pages
volume_and_number = str_extract(journal_to_clean, extract_volume_and_number), # extracting standard volument and number: X (X)
journal_to_clean = str_remove(journal_to_clean, " (p)?p\\. ([A-Z])?\\d+(-([A-Z])?\\d+)?"), # clean from extracted pages
journal_to_clean = str_remove(journal_to_clean, "( |^)?\\d+ \\(\\d+(-\\d+)?\\)"), # clean from extracted volume and number
volume_and_number = ifelse(is.na(volume_and_number), str_extract(journal_to_clean, "(?<= )([A-Z])?\\d+(-\\d+)?"), volume_and_number), # extract remaining numbers
journal_to_clean = str_remove(journal_to_clean, " ([A-Z])?\\d+(-\\d+)?"), # clean from remaining numbers
journal = str_remove_all(journal_to_clean, "^[:punct:]+( )?[:punct:]+( )?|(?<=,( )?)[:punct:]+( )?([:punct:])?|[:punct:]( )?[:punct:]+( )?$"), # extract journal info by removing inappropriate punctuations
first_page = str_extract(pages, "\\d+"),
volume = str_extract(volume_and_number, "\\d+"),
issue = str_extract(volume_and_number, "(?<=\\()\\d+(?=\\))"),
publisher = ifelse(is.na(first_page) & is.na(volume) & is.na(issue) & ! str_detect(journal, "(W|w)orking (P|p)?aper"), journal, NA),
book_title = ifelse(str_detect(journal, " (E|e)d(s)?\\.| (E|e)dite(d|urs)? "), journal, NA), # Incollection article: Title of the book here
book_title = str_extract(book_title, "[A-z ]+(?=,)"), # keeping only the title of the book
publisher = ifelse(!is.na(book_title), NA, publisher), # if we have an incollection article, that's not a book, so no publisher
journal = ifelse(!is.na(book_title) | ! is.na(publisher), NA, journal), # removing journal as what we have is a book
publisher = ifelse(is.na(publisher) & str_detect(journal, "(W|w)orking (P|p)?aper"), journal, publisher), # adding working paper publisher information in publisher column
journal = ifelse(str_detect(journal, "(W|w)orking (P|p)?aper"), "Working Paper", journal))
cleaned_articles <- cleaning_articles %>%
select(citing_id, id_ref, authors, year, title, journal, volume, issue, pages, first_page, book_title, publisher, references)
Nous faisons maintenant de même avec les références restantes qui sont moins nombreuses mais qui sont aussi moins faciles à nettoyer, du fait que le titre n’est pas clairement séparé des autres informations (revue ou éditeur).
cleaning_non_articles <- cleaning_references %>%
filter(is_article == FALSE) %>%
mutate(remaining_ref = str_remove(remaining_ref, "\\(\\d{4}\\)(,)? "),
title = str_extract(remaining_ref, ".*(?=, ,)"),
pages = str_extract(remaining_ref, "(?<= (p)?p\\. )([A-Z])?\\d+(-([A-Z])?\\d+)?"), # extracting pages
volume_and_number = str_extract(remaining_ref, "(?<=( |^)?)\\d+ \\(\\d+(-\\d+)?\\)"), # extracting standard volument and number: X (X)
remaining_ref = str_remove(remaining_ref, " (p)?p\\. ([A-Z])?\\d+(-([A-Z])?\\d+)?"), # clean from extracted pages
remaining_ref = str_remove_all(remaining_ref, ".*, ,"), # clean dates and already extracted titles
remaining_ref = str_remove(remaining_ref, "( |^)?\\d+ \\(\\d+(-\\d+)?\\)"), # clean from extracted volume and number
volume_and_number = ifelse(is.na(volume_and_number), str_extract(remaining_ref, "(?<= )([A-Z])?\\d+(-\\d+)?"), volume_and_number), # extract remaining numbers
remaining_ref = str_remove(remaining_ref, " ([A-Z])?\\d+(-\\d+)?"), # clean from remaining numbers
journal = ifelse(str_detect(remaining_ref, "(W|w)orking (P|p)aper"), "Working Paper", NA),
journal = ifelse(str_detect(remaining_ref, "(M|m)anuscript"), "Manuscript", journal),
journal = ifelse(str_detect(remaining_ref, "(M|m)imeo"), "Mimeo", journal),
publisher = ifelse(is.na(journal), remaining_ref, NA) %>% str_trim("both"),
first_page = str_extract(pages, "\\d+"),
volume = str_extract(volume_and_number, "\\d+"),
issue = str_extract(volume_and_number, "(?<=\\()\\d+(?=\\))"),
book_title = NA) # to be symetric with "cleaned_articles"
cleaned_non_articles <- cleaning_non_articles %>%
select(citing_id, id_ref, authors, year, title, journal, volume, issue, pages, first_page, book_title, publisher, references)
Nous fusionnons les deux data.frames et :
- nous normalisons et nettoyons le nom des auteurs afin de faciliter la mise en correspondance des références par la suite ;
- nous extrayons des informations utiles telles que DOI et PII.
# merging the two files.
cleaned_ref <- rbind(cleaned_articles, cleaned_non_articles)
#' Now we have all the references, we can do a bit of cleaning on the authors name,
#' and extract useful information, like DOI, for matching later.
cleaned_ref <- cleaned_ref %>%
mutate(authors = str_remove(authors, " Jr\\."), # standardising authors name to favour matching later
authors = str_remove(authors, "^\\(\\d{4}\\)(\\.)?( )?"),
authors = str_remove(authors, "^, "),
authors = ifelse(is.na(authors), str_extract(references, ".*[:upper:]\\.(?= \\d{4})"), authors), # specific case
journal = str_remove(journal, "[:punct:]$"), # remove unnecessary punctuations at the end
doi = str_extract(references, "(?<=DOI(:)? ).*|(?<=\\/doi\\.org\\/).*"),
pii = str_extract(doi, "(?<=PII ).*"),
doi = str_remove(doi, ",.*"), # cleaning doi
pii = str_remove(pii, ",.*"), # cleaning pii
)
knitr::kable(head(cleaned_ref, n = 3))
| citing_id | id_ref | authors | year | title | journal | volume | issue | pages | first_page | book_title | publisher | references | doi | pii |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A1 | 1 | Bernanke, B., Gertler, M., Gilchrist, S. | 1998 | Financial accelerator in a quantitative business cycle framework, NBER Working Paper No. W6455 | NA | NA | NA | NA | NA | NA | NBER | Bernanke, B., Gertler, M., Gilchrist, S., Financial accelerator in a quantitative business cycle framework, NBER Working Paper No. W6455 (1998), NBER | NA | NA |
| A1 | 2 | Calvo, G. | 1983 | Staggered prices in a utility maximizing framework | Journal of Monetary Economics | 12 | NA | 383-398 | 383 | NA | NA | Calvo, G., Staggered prices in a utility maximizing framework (1983) Journal of Monetary Economics, 12, pp. 383-398 | NA | NA |
| A1 | 3 | Christensen, I., Dib, A. | 2005 | Monetary policy in an estimated DSGE model with a financial accelerator | Computing in Economics and Finance | NA | NA | 314 | 314 | NA | NA | Christensen, I., Dib, A., Monetary policy in an estimated DSGE model with a financial accelerator (2005) Computing in Economics and Finance, p. 314 | NA | NA |
En pratique, si vous visez un travail quantitatif sérieux, un nettoyage plus poussé sera nécessaire pour éliminer les petites erreurs. Comme souvent dans ce type de nettoyage automatisé, 95% du nettoyage peut être fait avec quelques lignes de code, et le reste implique beaucoup plus de travail. Je n’irai pas plus loin car il s’agit d’un simple tutoriel.
Correspondance des références
Ce que nous devons faire maintenant, c’est trouver quelles références sont les mêmes, afin de leur donner un identifiant unique. Le compromis consiste à faire correspondre autant de vrais positifs que possible (références identiques) tout en évitant de faire correspondre des faux positifs, c’est-à-dire des références qui ont des informations en commun, mais qui ne sont en fait pas les mêmes références. Par exemple, l’appariement sur la base du nom des auteurs et de l’année de publication est trop large, car ces auteurs peuvent avoir publié plusieurs articles au cours de la même année. Voici plusieurs façons d’identifier une référence commune qui comportent très peu de risques d’appariement de références différentes :
- même nom de famille du ou des premiers auteurs, même année, même volume et même page (ce sont les plus sûrs) : appelons-les
fayvp&ayvp; - même revue, même volume, même numéro et même première page :
jvip; - même auteur, même année et même titre :
ayt; - même titre, même année et même première page :
typ; - même Doi ou PII [j’ai peut-être oublié quelques combinaisons utiles].
Nous extrayons le nom de famille du premier auteur pour favoriser l’appariement car il y a plus de possibilités de petites différences pour plusieurs auteurs qui nous empêcheraient d’apparier des références similaires5.
cleaned_ref <- cleaned_ref %>%
mutate(first_author = str_extract(authors, "^[[:alpha:]+[']?[ -]?]+, ([A-Z]\\.[ -]?)?([A-Z]\\.[ -]?)?([A-Z]\\.)?[A-Z]\\.(?=(,|$))"),
first_author_surname = str_extract(first_author, ".*(?=,)"),
across(.cols = c("authors", "first_author", "journal", "title"), ~toupper(.)))
Pour chaque type d’appariement, nous donnons un nouvel identifiant aux références appariées, en donnant le id_ref des premières références appariées. A la fin, nous comparons tous les nouveaux identifiants créés avec toutes les méthodes d’appariement, et nous prenons l’identifiant le plus petit.
matching_ref <- function(data, id_ref, ..., col_name){
match <- data %>%
group_by(...) %>%
mutate(new_id = min({{id_ref}})) %>%
drop_na(...) %>%
ungroup() %>%
select({{id_ref}}, new_id) %>%
rename_with(~ paste0(col_name, "_new_id"), .cols = new_id)
data <- data %>%
left_join(match)
}
identifying_ref <- cleaned_ref %>%
matching_ref(id_ref, first_author_surname, year, title, col_name = "fayt") %>%
matching_ref(id_ref, journal, volume, issue, first_page, col_name = "jvip") %>%
matching_ref(id_ref, authors, year, volume, first_page, col_name = "ayvp") %>%
matching_ref(id_ref, first_author_surname, year, volume, first_page, col_name = "fayvp") %>%
matching_ref(id_ref, title, year, first_page, col_name = "typ") %>%
matching_ref(id_ref, pii, col_name = "pii") %>%
matching_ref(id_ref, doi, col_name = "doi")
Nous avons maintenant notre tableau de citations directes reliant les articles citants aux références. Nous avons autant de lignes que le nombre de citations d’articles.
direct_citation <- identifying_ref %>%
mutate(new_id_ref = select(., ends_with("new_id")) %>% reduce(pmin, na.rm = TRUE),
new_id_ref = ifelse(is.na(new_id_ref), id_ref, new_id_ref)) %>%
relocate(new_id_ref, .after = citing_id) %>%
select(-id_ref & ! ends_with("new_id"))
knitr::kable(head(filter(direct_citation, new_id_ref == 2), n = 4))
| citing_id | new_id_ref | authors | year | title | journal | volume | issue | pages | first_page | book_title | publisher | references | doi | pii | first_author | first_author_surname |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A1 | 2 | CALVO, G. | 1983 | STAGGERED PRICES IN A UTILITY MAXIMIZING FRAMEWORK | JOURNAL OF MONETARY ECONOMICS | 12 | NA | 383-398 | 383 | NA | NA | Calvo, G., Staggered prices in a utility maximizing framework (1983) Journal of Monetary Economics, 12, pp. 383-398 | NA | NA | CALVO, G. | Calvo |
| A3 | 2 | CALVO, G. | 1983 | STAGGERED PRICES IN A UTILITY-MAXIMIZING FRAMEWORK | JOURNAL OF MONETARY ECONOMICS | 12 | 3 | 383-398 | 383 | NA | NA | Calvo, G., Staggered Prices in a Utility-Maximizing Framework (1983) Journal of Monetary Economics, 12 (3), pp. 383-398 | NA | NA | CALVO, G. | Calvo |
| A18 | 2 | CALVO, G. | 1983 | STAGGERED PRICES IN A UTILITY MAXIMIZING FRAMEWORK | JOURNAL OF MONETARY ECONOMICS | 12 | NA | 383-398 | 383 | NA | NA | Calvo, G., Staggered prices in a utility maximizing framework (1983) Journal of Monetary Economics, 12, pp. 383-398 | NA | NA | CALVO, G. | Calvo |
| A20 | 2 | CALVO, G.A. | 1983 | STAGGERED PRICES IN A UTILITY-MAXIMIZING FRAMEWORK | JOURNAL OF MONETARY ECONOMICS | 12 | 3 | 383-398 | 383 | NA | NA | Calvo, G.A., Staggered prices in a utility-maximizing framework (1983) Journal of Monetary Economics, 12 (3), pp. 383-398 | NA | NA | CALVO, G.A. | Calvo |
Nous pouvons extraire la liste de toutes les références citées. Nous avons autant de lignes que de références citées par les articles citants (c’est-à-dire qu’une référence citée plusieurs fois n’est présente qu’une seule fois dans le tableau). Comme pour les références appariées, nous disposons d’informations différentes (dues au fait que les références ont été citées différemment selon les articles citants), nous prenons une ligne où l’information semble la plus complète.
important_info <- c("authors",
"year",
"title",
"journal",
"volume",
"issue",
"pages",
"book_title",
"publisher")
references <- direct_citation %>%
mutate(nb_na = rowSums(!is.na(select(., all_of(important_info))))) %>%
group_by(new_id_ref) %>%
slice_max(order_by = nb_na, n = 1, with_ties = FALSE) %>%
select(-citing_id) %>%
unique
knitr::kable(head(references, n = 4))
| new_id_ref | authors | year | title | journal | volume | issue | pages | first_page | book_title | publisher | references | doi | pii | first_author | first_author_surname | nb_na |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | BERNANKE, B., GERTLER, M., GILCHRIST, S. | 1998 | FINANCIAL ACCELERATOR IN A QUANTITATIVE BUSINESS CYCLE FRAMEWORK, NBER WORKING PAPER NO. W6455 | NA | NA | NA | NA | NA | NA | NBER | Bernanke, B., Gertler, M., Gilchrist, S., Financial accelerator in a quantitative business cycle framework, NBER Working Paper No. W6455 (1998), NBER | NA | NA | BERNANKE, B. | Bernanke | 4 |
| 2 | CALVO, G. | 1983 | STAGGERED PRICES IN A UTILITY-MAXIMIZING FRAMEWORK | JOURNAL OF MONETARY ECONOMICS | 12 | 3 | 383-398 | 383 | NA | NA | Calvo, G., Staggered Prices in a Utility-Maximizing Framework (1983) Journal of Monetary Economics, 12 (3), pp. 383-398 | NA | NA | CALVO, G. | Calvo | 7 |
| 3 | CHRISTENSEN, I., DIB, A. | 2005 | MONETARY POLICY IN AN ESTIMATED DSGE MODEL WITH A FINANCIAL ACCELERATOR | COMPUTING IN ECONOMICS AND FINANCE | NA | NA | 314 | 314 | NA | NA | Christensen, I., Dib, A., Monetary policy in an estimated DSGE model with a financial accelerator (2005) Computing in Economics and Finance, p. 314 | NA | NA | CHRISTENSEN, I. | Christensen | 5 |
| 4 | DIXIT, A., STIGLITZ, J. | 1977 | MONOPOLISTIC COMPETITION AND OPTIMUM PRODUCT DIVERSITY | AMERICAN ECONOMIC REVIEW | 67 | 3 | 297-308 | 297 | NA | NA | Dixit, A., Stiglitz, J., Monopolistic competition and optimum product diversity (1977) American Economic Review, 67 (3), pp. 297-308 | NA | NA | DIXIT, A. | Dixit | 7 |
A partir des 105313 citations initiales, nous obtenons 52577 références différentes, avec 10631 cité au moins deux fois.
Nous supprimons également la colonne references du data.frame initial car les références citées sont maintenant regroupées dans direct_citation.
corpus <- scopus_search %>%
select(-references)
J’imagine qu’après tant d’étapes, vous avez hâte de consulter ces données sur les modèles DSGE. Mais vous devrez attendre, car la sous-section suivante vous présente une autre méthode pour nettoyer (sans vous nettoyer vous-même) les références. Vous pouvez aussi aller directement à la [dernière section] (#exploring-the-dsge-literature) qui explore les données nettoyées.
Méthode alternative : anystyle
anystyle est un excellent analyseur de références qui s’appuie sur des heuristiques d’apprentissage automatique. Il dispose d’une version en ligne dans laquelle vous pouvez placer le texte pour lequel vous souhaitez identifier des références. Cependant, nous utiliserons la ligne de commande afin d’identifier plus de références que ce que le site en ligne permet (10 000 alors que nous avons 105313 références brutes).
Anystyle peut être installé en tant que RubyGem. Vous devez donc installer Ruby (ici pour Windows) et ensuite installer anystyle en utilisant la ligne de commande : gem install anystyle (voir plus d’informations ici). Comme vous devez utiliser l’interface en ligne de commande, vous devez aussi installer anystyle-cli : gem install anystyle-cli.
Un lecteur effrayé : “Quoi, quoi ? Attendez ! Ligne de commande vous avez dit ?”
Oh, oui. Et c’est une bonne occasion de vous renvoyer à ces excellents tutoriels du Programming Historian Website : un pour la ligne de commande bash et un pour la ligne de commande Windows Powershell. Et ce sera l’occasion d’utiliser un peu de Rstudio Terminal pour entrer les commandes.
Une fois que vous avez installé anystyle, vous devez sauvegarder toutes les références (avec une référence par ligne) dans un fichier .txt.
ref_text <- paste0(references_extract$references, collapse = "\\\n")
name_file <- "ref_in_text.txt"
write_file(ref_text,
here(scopus_path,
name_file))
Pour créer la commande anystyle, vous devez nommer le dépôt où vous enverrez le .bib créé par anystyle à partir de votre .txt.
destination_anystyle <- "anystyle_cleaned"
directory_command <- paste0("cd ", scopus_path)
anystyle_command <- paste0("anystyle -f bib parse ",
name_file,
" ",
destination_anystyle)
Pour utiliser anystyle, vous devez utiliser la ligne de commande du terminal. Vous devez d’abord définir le chemin où se trouve le .txt (qui est ici le scopus_path) : cd le_chemin_où_est_le_.txt.
Ensuite, vous copiez et collez la commande anystyle dans le terminal, qui est ici :
anystyle -f bib parse ref_in_text.txt anystyle_cleaned. Avec un peu de chance, cela fonctionnera et vous n’aurez qu’à attendre la création du .bib (cela a pris environ 10 minutes sur mon ordinateur portable)6.
…en attendant…
Une fois que nous avons notre .bib, nous le transformons en data frame grâce au package bib2df.
options(encoding = "UTF-8")
bib_ref <- bib2df(here(scopus_path,
destination_anystyle,
"ref_in_text.bib"))
bib_ref <- bib_ref %>%
janitor::clean_names() %>%
select_if(~!all(is.na(.))) %>% # removing all empty columns
mutate(id_ref = 1:n()) %>%
select(-c(translator, citation_number, arxiv, director, source))
knitr::kable(head(bib_ref))
| category | bibtexkey | address | author | booktitle | edition | editor | institution | journal | note | number | pages | publisher | school | series | title | type | volume | date | issue | url | isbn | doi | id_ref |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ARTICLE | bernanke1998a | NA | Bernanke, B. , Gertler, M. , Gilchrist, S. | NA | NA | NA | NA | NBER Working Paper | NA | W6455 | NA | NA | NA | NA | Financial accelerator in a quantitative business cycle framework | NA | 1998 | NA | NA | NA | NA | 1 | |
| ARTICLE | calvo-a | NA | Calvo, G. | NA | NA | NA | NA | Journal of Monetary Economics | NA | NA | 383–398 | NA | NA | NA | Staggered prices in a utility maximizing framework (1983 | NA | 12 | NA | NA | NA | NA | NA | 2 |
| ARTICLE | christensen-a | NA | Christensen, I., Dib, A. | NA | NA | NA | NA | Computing in Economics and Finance | NA | NA | 314 | NA | NA | NA | Monetary policy in an estimated DSGE model with a financial accelerator (2005 | NA | NA | NA | NA | NA | NA | NA | 3 |
| ARTICLE | dixit-a | NA | Dixit, A. , Stiglitz, J. | NA | NA | NA | NA | American Economic Review | NA | 3 | 297–308 | NA | NA | NA | Monopolistic competition and optimum product diversity (1977 | NA | 67 | NA | NA | NA | NA | NA | 4 |
| BOOK | gerali2010a | Banka D’Italia, Rome | Gerali, A. , Neri, S. , Sessa, L. , Signoretti, F. | NA | NA | NA | NA | NA | NA | NA | 107–141 | , Working paper | NA | NA | Credit and banking in a DSGE model | NA | 42 | 2010 | 6 | NA | NA | NA | 5 |
| INCOLLECTION | goodfriend-a | NA | Goodfriend, M., McCallum, B. | NBER Working Paper Series No. 13207, NBER\ | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | Banking and interest rates in monetary policy analysis: A quantitative exploration (2007 | NA | NA | NA | NA | NA | NA | NA | 6 |
Pour l’instant, il y a une limitation majeure à cette méthode (qui est très probablement liée à mon manque de maîtrise d’anystyle et de ruby) : le résultat est une liste de références uniques. En d’autres termes, anystyle fusionne les références qui sont similaires. Cela signifie que je dois trouver un moyen de construire un lien entre les références originales data.frame et le data.frame construit sur le .bib.7.
Idéalement, vous pouvez nettoyer un peu le résultat. Anystyle est assez bon (et clairement meilleur que moi) pour identifier les types de références, et donc pour extraire le titre du livre pour les chapitres dans les livres et les éditeurs. Il est assez efficace pour extraire les auteurs, et aussi les titres même si j’ai vu beaucoup d’erreurs (mais relativement à nettoyer, car la plupart du temps c’est l’année qui a été mise avec le titre). Cependant, j’ai aussi vu beaucoup d’erreurs pour les revues (nom incomplet) qui ne sont pas si faciles à corriger. Si vous souhaitez nettoyer vos données de références autant que possible, la meilleure chose à faire est peut-être de combiner l’approche de nettoyage par codage utilisée ci-dessus avec la méthode anystyle, et de compléter les informations manquantes avec l’une ou l’autre méthode.
Exploration de la littérature sur les DSGE
Maintenant que nous disposons de nos données bibliographiques, la première chose que nous pouvons faire est d’examiner les références les plus citées dans notre corpus.
direct_citation %>%
add_count(new_id_ref) %>%
select(new_id_ref, n) %>%
unique() %>%
slice_max(n, n = 10) %>%
left_join(select(references, new_id_ref, references)) %>%
select(references, n) %>%
knitr::kable()
| references | n |
|---|---|
| Smets, F., Wouters, R., Shocks and frictions in US business cycles. a Bayesian DSGE approach (2007) American Economic Review, 97 (3), pp. 586-606 | 780 |
| Calvo, G., Staggered Prices in a Utility-Maximizing Framework (1983) Journal of Monetary Economics, 12 (3), pp. 383-398 | 702 |
| Christiano, L.J., Eichenbaum, M., Evans, C.L., Nominal rigidities and the dynamic effects of a shock to monetary policy (2005) Journal of Political Economy, 113 (1), pp. 1-45. , http://ideas.repec.org/a/ucp/jpolec/v113y2005i1p1-45.html | 640 |
| Smets, F., Wouters, R., An Estimated Dynamic Stochastic General Equilibrium Model of the Euro Area (2003) Journal of the European Economic Association, 1 (5), pp. 1123-1175 | 610 |
| Bernanke, B., Gertler, M., Gilchrist, S., The financial accelerator in a quantitative business cycle framework (1999) NBER Working Papers Series, 1 (3), pp. 1341-1393. , Elsevier Science B.V, (chap. 21), Handbook of Macroeconomics | 331 |
| An, S., Schorfheide, F., Bayesian Analysis of DSGE Models (2007) Econometric Reviews, 26 (4), pp. 113-172 | 311 |
| Taylor, J., Discretion versus policy rules in practice (1993) Carnegie-Rochester Conference Series on Public Policy, 39 (0), pp. 195-214 | 269 |
| Iacoviello, M., House prices, borrowing constraints, and monetary policy in the business cycle (2005) American Economic Review, 95 (3), pp. 739-764 | 243 |
| Kydland, F.E., Prescott, E.C., Time to build and aggregate fluctuations (1982) Econometrica, 50 (6), pp. 1345-1370 | 234 |
| Schmitt-Grohe, S., Uribe, M., Closing small open economy models (2003) Journal of International Economics, 61 (1), pp. 163-185 | 228 |
Comme nous disposons des affiliations, nous pouvons essayer de voir quelles sont les principales références pour les économistes basés dans différents pays :
direct_citation %>%
left_join(select(scopus_affiliations, citing_id, country)) %>%
unique() %>%
group_by(country) %>%
mutate(nb_article = n()) %>%
filter(nb_article > 5000) %>% # we keep only countries with 5000 articles
add_count(new_id_ref) %>%
select(new_id_ref, n) %>%
unique() %>%
slice_max(n, n = 8) %>%
left_join(select(references, new_id_ref, references)) %>%
select(references, n) %>%
mutate(label = str_extract(references, ".*\\(\\d{4}\\)") %>%
str_wrap(30),
label = tidytext::reorder_within(label, n, country)) %>%
ggplot(aes(n, label, fill = country)) +
geom_col(show.legend = FALSE) +
facet_wrap(~country, ncol = 3, scales = "free") +
tidytext::scale_y_reordered() +
labs(x = "Number of citations", y = NULL) +
theme_classic(base_size = 10)
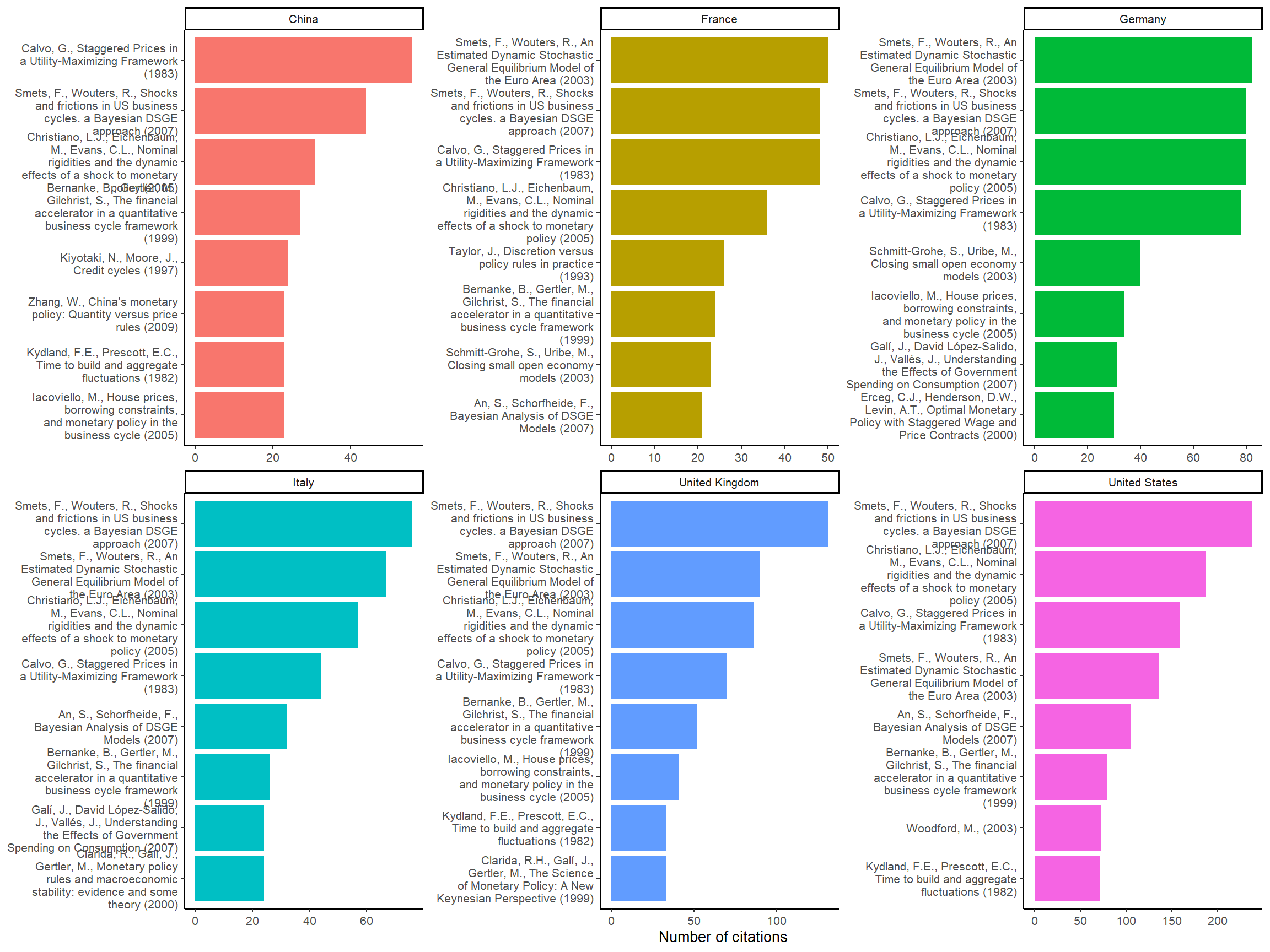
Figure 4: Most cited references per countries
En utilisant les affiliations, nous pouvons observer un modèle de préférence régionale : dans les pays européens, les économistes ont tendance à citer davantage Smets and Wouters (2003) (la source du modèle DSGE de la Banque centrale européenne) que Christiano, Eichenbaum, and Evans (2005), alors que l’inverse est vrai aux États-Unis (Smets and Wouters 2007 est sur des données américaines). Nous pouvons également remarquer que Kydland and Prescott (1982) est moins populaire en Europe continentale.
Analyse des co-citations bibliographiques
Pour conclure ce (long) tutoriel, nous pouvons construire un réseau de co-citation : les références que nous avons appariées sont les noeuds du réseau, et elles sont reliées entre elles en fonction du nombre de fois qu’elles sont citées ensemble (ou en d’autres termes, le nombre de fois qu’elles sont ensemble dans une bibliographie). Nous utilisons la fonction biblio_cocitation du package biblionetwork. L’arête entre deux nœuds est pondérée en fonction du nombre total de fois où chaque référence a été citée dans l’ensemble du corpus (voir ici pour plus de détails).
citations <- direct_citation %>%
add_count(new_id_ref) %>%
select(new_id_ref, n) %>%
unique
references_filtered <- references %>%
left_join(citations) %>%
filter(n >= 5)
edges <- biblionetwork::biblio_cocitation(filter(direct_citation, new_id_ref %in% references_filtered$new_id_ref),
"citing_id",
"new_id_ref",
weight_threshold = 3)
edges
## Key: <Source>
## from to weight Source Target
## <char> <char> <num> <int> <int>
## 1: 2 4 0.2354379 2 4
## 2: 2 5 0.1244966 2 5
## 3: 2 6 0.0518193 2 6
## 4: 2 7 0.1623352 2 7
## 5: 2 14 0.1349191 2 14
## ---
## 42657: 64761 64841 0.4045199 64761 64841
## 42658: 64841 64936 0.3481553 64841 64936
## 42659: 66416 68931 0.7453560 66416 68931
## 42660: 66416 68935 0.5039526 66416 68935
## 42661: 68931 68935 0.6761234 68931 68935
Nous pouvons alors prendre notre corpus et ces arêtes pour créer un réseau/graphique grâce à tidygraph (Pedersen 2020) et networkflow. Je n’entre pas dans les détails ici car ce n’est pas le but de ce tutoriel et que les différentes étapes sont expliquées sur le site de networkflow.
graph <- tbl_main_component(nodes = references_filtered,
edges = edges,
directed = FALSE)
graph
## # A tbl_graph: 2836 nodes and 42661 edges
## #
## # An undirected simple graph with 1 component
## #
## # A tibble: 2,836 × 18
## Id authors year title journal volume issue pages first_page book_title
## <int> <chr> <int> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 2 CALVO, G. 1983 "STA… JOURNA… 12 3 383-… 383 <NA>
## 2 4 DIXIT, A.,… 1977 "MON… AMERIC… 67 3 297-… 297 <NA>
## 3 5 GERALI, A.… 2010 "CRE… WORKIN… 42 6 107-… 107 <NA>
## 4 6 GOODFRIEND… 2007 "BAN… JOURNA… 54 5 1480… 1480 <NA>
## 5 7 IACOVIELLO… 2005 "HOU… AMERIC… 95 3 739-… 739 <NA>
## 6 14 ROTEMBERG,… 1982 "MON… REVIEW… 49 4 517-… 517 <NA>
## # ℹ 2,830 more rows
## # ℹ 8 more variables: publisher <chr>, references <chr>, doi <chr>, pii <chr>,
## # first_author <chr>, first_author_surname <chr>, nb_na <dbl>, n <int>
## #
## # A tibble: 42,661 × 5
## from to weight Source Target
## <int> <int> <dbl> <int> <int>
## 1 1 2 0.235 2 4
## 2 1 3 0.124 2 5
## 3 1 4 0.0518 2 6
## # ℹ 42,658 more rows
set.seed(1234)
graph <- leiden_workflow(graph) # identifying clusters of nodes
nb_communities <- graph %>%
activate(nodes) %>%
as_tibble %>%
select(Com_ID) %>%
unique %>%
nrow
palette <- scico::scico(n = nb_communities, palette = "hawaii") %>% # creating a color palette
sample()
graph <- community_colors(graph, palette, community_column = "Com_ID")
graph <- graph %>%
activate(nodes) %>%
mutate(size = n,# will be used for size of nodes
label = paste0(first_author_surname, "-", year))
graph <- community_names(graph,
ordering_column = "size",
naming = "label",
community_column = "Com_ID")
graph <- vite::complete_forceatlas2(graph,
first.iter = 10000)
top_nodes <- top_nodes(graph,
ordering_column = "size",
top_n = 15,
top_n_per_com = 2,
biggest_community = TRUE,
community_threshold = 0.02)
community_labels <- community_labels(graph,
community_name_column = "Community_name",
community_size_column = "Size_com",
biggest_community = TRUE,
community_threshold = 0.02)
Un réseau de co-citations permet d’observer quelles sont les principales influences d’un domaine de recherche. Au centre du réseau, nous trouvons les références les plus citées. Sur les bords du graphe, on trouve des communautés spécifiques qui influencent différentes parties de la littérature sur le DSGE. Ici, la taille des nœuds dépend du nombre de fois qu’ils sont cités dans notre corpus.
graph <- graph %>%
activate(edges) %>%
filter(weight > 0.05)
ggraph(graph, "manual", x = x, y = y) +
geom_edge_arc0(aes(color = color_edges, width = weight), alpha = 0.4, strength = 0.2, show.legend = FALSE) +
scale_edge_width_continuous(range = c(0.1,2)) +
scale_edge_colour_identity() +
geom_node_point(aes(x=x, y=y, size = size, fill = color), pch = 21, alpha = 0.7, show.legend = FALSE) +
scale_size_continuous(range = c(0.3,16)) +
scale_fill_identity() +
ggnewscale::new_scale("size") +
ggrepel::geom_text_repel(data = top_nodes, aes(x=x, y=y, label = Label), size = 2, fontface="bold", alpha = 1, point.padding=NA, show.legend = FALSE) +
ggrepel::geom_label_repel(data = community_labels, aes(x=x, y=y, label = Community_name, fill = color), size = 6, fontface="bold", alpha = 0.9, point.padding=NA, show.legend = FALSE) +
scale_size_continuous(range = c(0.5,5)) +
theme_void()
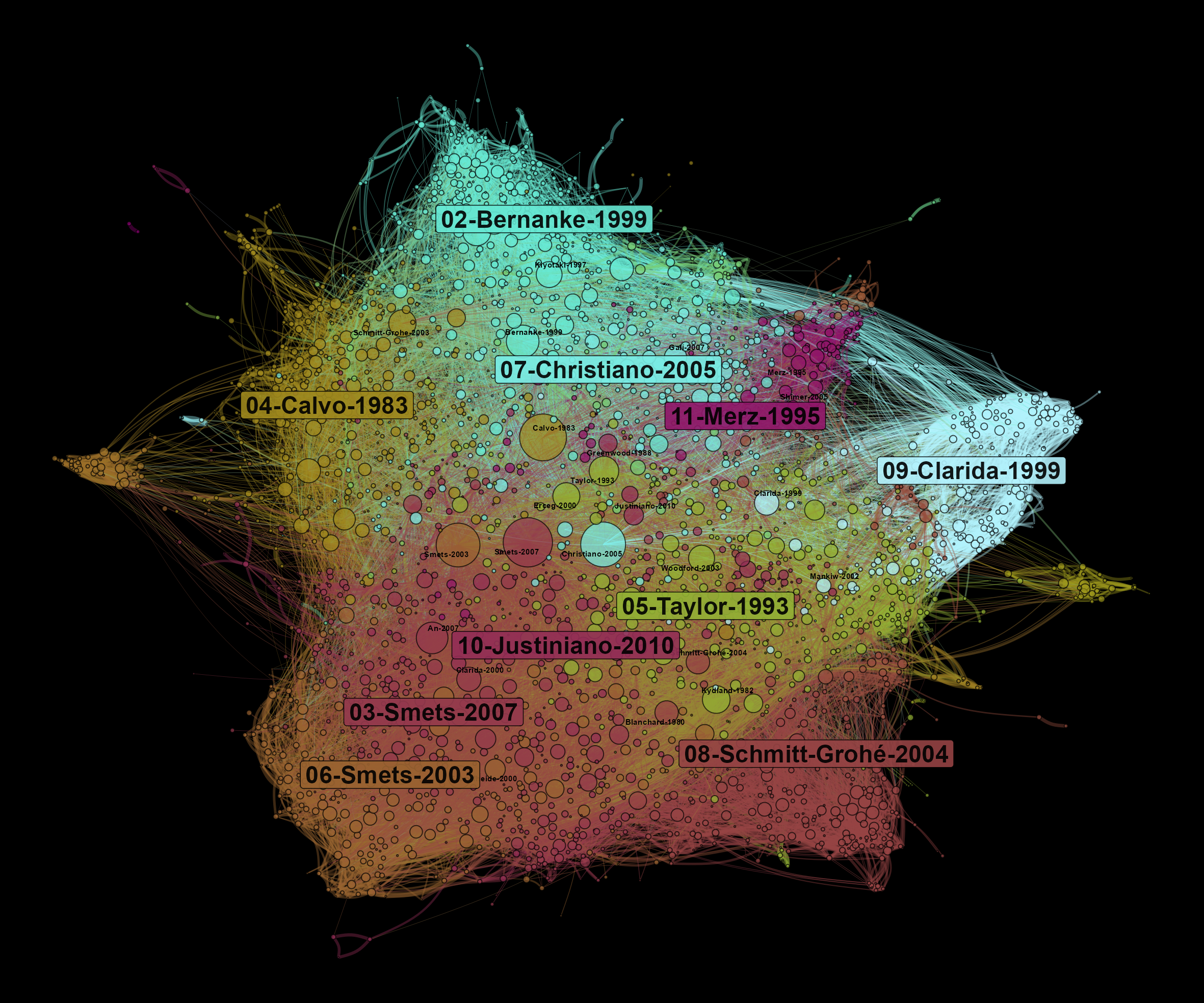
Figure 5: Bibliographic coupling network of articles using DSGE models
Terminons en observant quels sont les nœuds les plus cités dans chaque communauté. Nous constatons que la communauté 04 traite des questions internationales tandis que la 07 est liée aux questions de politique fiscale.
ragg::agg_png(here("content", "en", "post", "2022-01-31-extracting-biblio-data-1", "top-ref-country-1.png"),
width = 35,
height = 30,
units = "cm",
res = 200)
top_nodes(graph,
ordering_column = "size",
top_n_per_com = 6,
biggest_community = TRUE,
community_threshold = 0.04) %>%
select(Community_name, Label, title, n, color) %>%
mutate(label = paste0(Label, "-", title) %>%
str_wrap(34),
label = tidytext::reorder_within(label, n, Community_name)) %>%
ggplot(aes(n, label, fill = color)) +
geom_col(show.legend = FALSE) +
scale_fill_identity() +
facet_wrap(~Community_name, ncol = 3, scales = "free") +
tidytext::scale_y_reordered() +
labs(x = "Number of citations", y = NULL) +
theme_classic(base_size = 11)
invisible(dev.off())
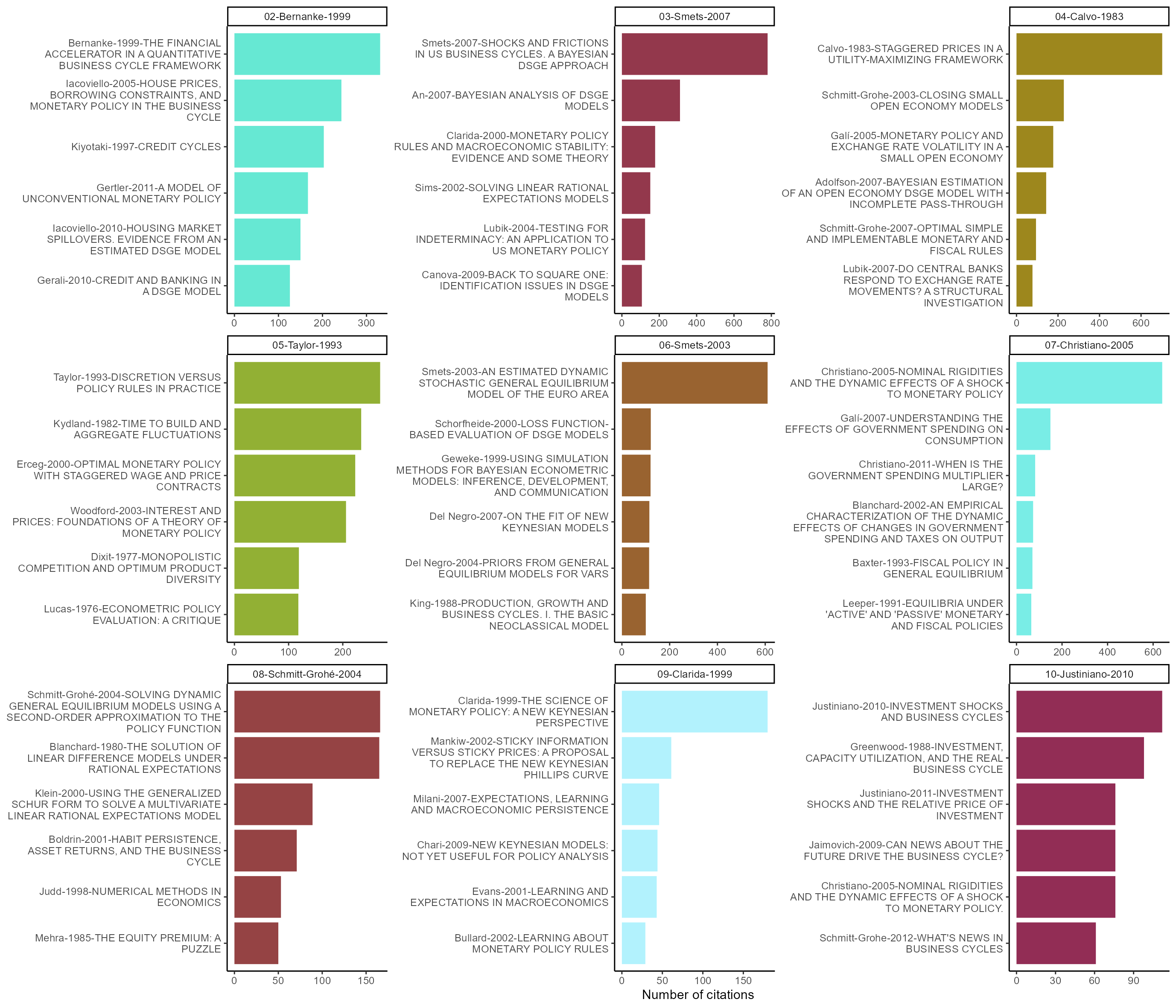
Figure 6: Most cited references per communities
Bibliographie
Christiano, Lawrence J., Martin Eichenbaum, and Charles L. Evans. 2005. “Nominal Rigidities and the Dynamic Effects of a Shock to Monetary Policy.” Journal of Political Economy 113 (1): 1–45.
De Vroey, Michel. 2016. A History of Macroeconomics from Keynes to Lucas and Beyond. Cambridge: Cambridge University Press.
Goutsmedt, Aurélien, François Claveau, and Alexandre Truc. 2021. Biblionetwork: Create Different Types of Bibliometric Networks.
Kydland, Finn E., and Edward C. Prescott. 1982. “Time to Build and Aggregate Fluctuations.” Econometrica: Journal of the Econometric Society 50 (6): 1345–70.
Pedersen, Thomas Lin. 2020. Tidygraph: A Tidy API for Graph Manipulation. https://CRAN.R-project.org/package=tidygraph.
Sergi, Francesco. 2020. “The Standard Narrative about DSGE Models in Central Banks’ Technical Reports.” The European Journal of the History of Economic Thought 27 (2): 163–93.
Smets, Frank, and Raf Wouters. 2003. “An Estimated Dynamic Stochastic General Equilibrium Model of the Euro Area.” Journal of the European Economic Association 1 (5): 1123–75.
Smets, Frank, and Rafael Wouters. 2007. “Shocks and Frictions in US Business Cycles: A Bayesian DSGE Approach.” American Economic Review 97 (3): 586–606.
Vines, David, and Samuel Wills. 2018. “The Rebuilding Macroeconomic Theory Project: An Analytical Assessment.” Oxford Review of Economic Policy 34 (1-2): 1–42. https://doi.org/10.1093/oxrep/grx062.
Wickham, Hadley. 2021. Tidyverse: Easily Install and Load the Tidyverse. https://CRAN.R-project.org/package=tidyverse.
Wickham, Hadley, Mara Averick, Jennifer Bryan, Winston Chang, Lucy D’Agostino McGowan, Romain François, Garrett Grolemund, et al. 2019. “Welcome to the tidyverse.” Journal of Open Source Software 4 (43): 1686. https://doi.org/10.21105/joss.01686.
Pour des discussions réflexives et historiques sur les modèles DSGE, voir De Vroey (2016, chapitre 20), Vines and Wills (2018) et Sergi (2020) ↩︎
Normalement, vous ne pourrez pas télécharger toutes les données en une seule extraction car il y a une limite de 2000 éléments, et vous devrez donc le faire en deux étapes. La plus simple consiste à filtrer par année ↩︎
Nous supprimons tous les
data.framescitationsqui sont vides (c’est à dire quand un article ne cite rien) ↩︎J’essaierai de vérifier cela pour un autre tutoriel sur les données bibliométriques, mais j’ai observé que j’ai obtenu moins de citations avec la méthode API qu’avec la méthode du site web de Scopus. Cela signifie peut-être que si Scopus n’a pas été en mesure de donner un identifiant à une référence (peut-être en raison de métadonnées pas suffisamment propres), la citation de cette référence est supprimée des données. Par conséquent, si notre méthode de nettoyage est bonne, nous pourrions être en mesure de conserver plus de citations et donc de conserver des références qui pourraient être exclues dans l’extraction des données des API ↩︎
La plupart des différences sont dues aux initiales des auteurs : certaines références n’en ont qu’une seule alors que d’autres ont deux initiales pour certains auteurs ↩︎
Au cas où vous voudriez en savoir plus sur les différentes commandes d’anystyle, consultez la [documentation API] (https://rubydoc.info/gems/anystyle) ↩︎
Comme je trouve qu’anystyle est un outil très intéressant, j’essaierai de travailler sur ce problème dans les mois à venir et de trouver une solution. Une façon simple de le faire serait peut-être d’enregistrer un
.txtpar article cité et de lancer anystyle pour tous les.txt. Nous aurons autant de.bibque d’articles et nous n’aurons plus qu’à lier lesdata.framesqui en résulteront ↩︎